Die Proteinreinigung ist der Prozess der Einführung von Nukleinsäuresequenzen, die durch genetische Technik, die für Proteine in Wirtszellen kodieren, durch genetische Technik, wodurch sie in großen Mengen exprimiert werden, und sie dann in vitro mit geeigneten Reinigungsmethoden reinigen, um Proteine mit hoher Reinheit, Aktivität und Ertrag zu erhalten . Proteinexpressionssysteme können in prokaryotische Systeme und eukaryotische Systeme kategorisiert werden.
Die Prinzipien und Methoden zur Induktion der Proteinexpression in prokaryotischen Systemen und zur Reinigung von Proteinen in vitro unter Verwendung von Affinitätschromatographie werden nachstehend beschrieben.
PRINZIP
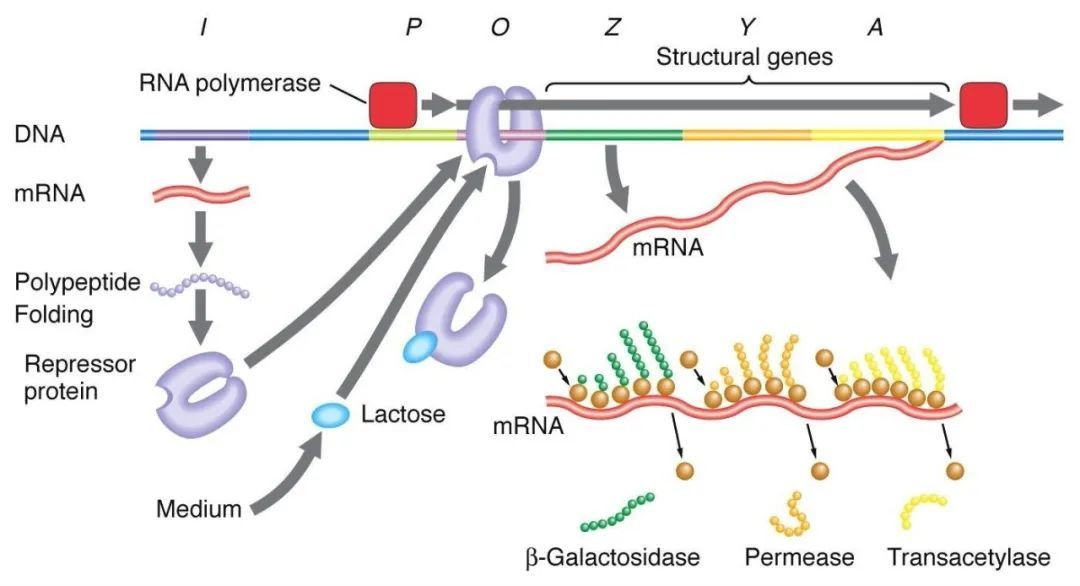
1 、 Negative Regulation des Laktosemanipulators:
In Abwesenheit von Lactose befindet sich der Lac -Manipulator in einem Zustand der Abschreckung, zu dem die I -Sequenz das Lac -Abschreckungsprotein an die O -Sequenz exprimiert, wodurch die Bindung von RNA -Polymerase an die P -Sequenz und die Hemmung der Transkriptionsinitiation verhindert wird. Wenn Lactose vorliegt, tritt Lactose in die Zelle ein und wird durch β-Galactosidase katalysiert und in Isolactose umgewandelt, was an das inhibitorische Protein bindet, was zu einer Konformationsänderung des Proteins führt und zur Dissoziation des inhibitorischen Proteins von der O-Sequenz führt, wodurch der Transkription initiiert wird.
Isopropylthiogalactosid (IPTG) wirkt auf die gleiche Weise wie Isogalactose und ist ein äußerst wirksamer Induktor, der nicht von Bakterien metabolisiert und sehr stabil ist und daher in Laboratorien häufig verwendet wird.
2 、 Auswahl der Hostdehnung:
(1) BL21 ist eines der am häufigsten verwendeten Stämme für die prokaryotische Expression, die hauptsächlich für die Expression ungiftiger Proteine mit E. coli-Polymerase geeignet ist, sodass es auf die Expression von prokaryotischen Systemen unter Verwendung von E. coli RNA-Polymerase wie TAC oder TRC (z. PGEX, PMAL-PLASMIDS) angewendet werden kann.
(2) BL21 (DE3) integriert das T7 -Phagen -RNA -Polymerase -Gen in die λ -Phagen -DE3 -Region auf dem Chromosom des BL21 -Stammes, das sowohl T7 -RNA -Polymerase als auch E. coli RNA -RNA -Polymerase exprimieren kann , und kann für die Expression von Plasmiden wie PET -Serien, PGEX, PMAL und so auf verwendet werden.
3, Proteinreinigungsmethoden:
. Die ersten, die aus den Molekülen herausgegeben werden, sind kleiner, je später die Elution die Elution.
. Wenn der pH -Wert geändert wird oder ein Puffer mit allmählich zunehmender Ionenstärke für die Elution verwendet wird, kann die gebundene Substanz mit den Ionen im Eluenten ausgetauscht und in die Lösung eluiert werden. Da unterschiedliche Substanzen mit der Ionenaustauschsäule unterschiedliche Ladungen und unterschiedliche Bindungsfähigkeiten aufweisen, ist auch die Reihenfolge der eluierten Lösung unterschiedlich.
, von hoch nach niedrig.
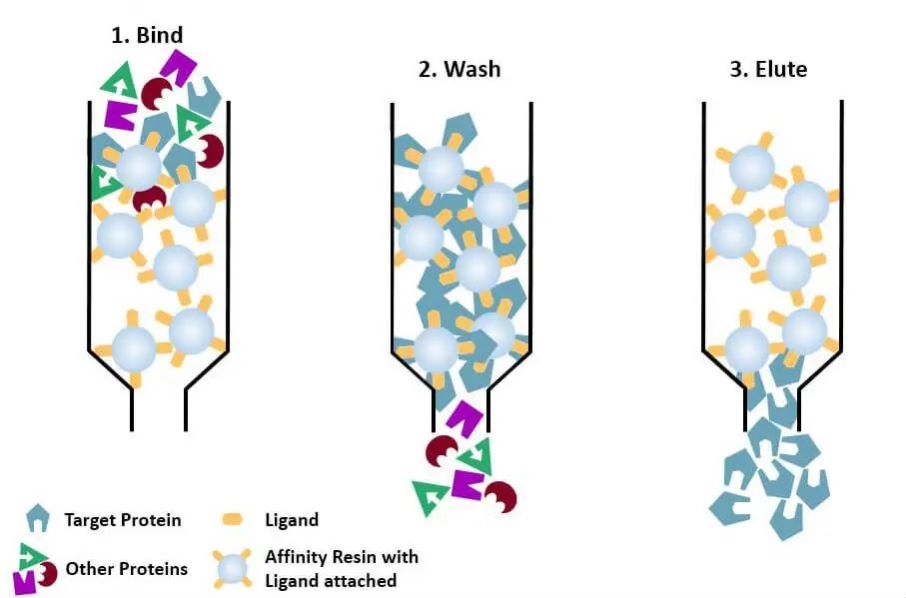
(4) Affinitätschromatographie: Der spezifische Ligand eines zu gersenen (oder markierten auf dem Protein markierten Protein) wird durch geeignete chemische Methoden kovalent an das Trägermolekül gebunden. Wenn die mit Affinitätsmedium gefüllte chromatographische Säule der Proteinmischung zugesetzt wird, ist das zu gersene Protein spezifisch an den Liganden gebunden, während die anderen Proteine nicht durch Waschen gebunden und entfernt werden und das spezifisch gebundene Protein mit einer Lösung freier entsprechender Ligand ausgelöst werden kann. Die spezifisch gebundenen Proteine können mit einer Lösung, die den freien entsprechenden Liganden enthält, eluiert werden.
4 、 Auswahl der Proteinreinigungsbezeichnungen:
Die Eigenschaften verschiedener Etiketten sind wie folgt ↓
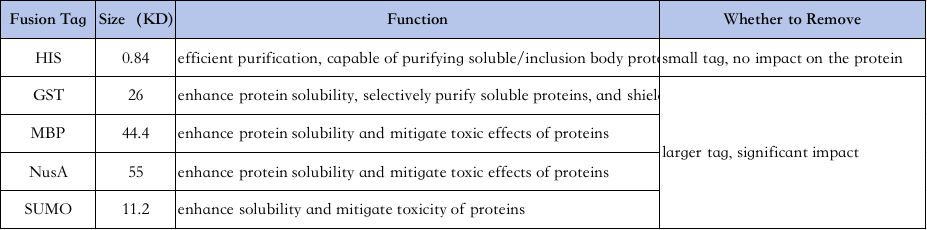
His-tag purification: His-tag is one of the most commonly used tags, where 6-10 histidine are added to the amino or carboxyl terminus of the protein, and the target protein is purified by its ability to bind tightly to Ni2+ chelating columns under normal or denaturing conditions (eg, 8M urea), and then eluted with imidazole (which competes for binding to nickel ions in the bead wells).
Instrumente und Verbrauchsmaterialien
Laborreagenzien:
Iptg; LB Medium;
Lysepuffer: 20 mm Tris (7,9), 500 mm NaCl, 10 mm Imidazol;
Waschpuffer: 20 mm Tris (7,9), 500 mm NaCl, 20 mm Imidazol;
Elutionspuffer: 20 mm Tris (7,9), 200 mm NaCl, 300 mm Imidazol ( Imidazolkonzentration kann gemäß der Elutionseffizienz eingestellt werden );
Dialyselösung: 20 mM Tris (7,9), 50 mM NaCl, 10% Glycerin;
Kaomas Brilliant Blue Fleckenlösung; Entfärbung der Lösung
Instrumente:
Ni-NTA, Ultraschallbrecher, Chromatographie-Säule, Konzentrationsrohre

Verfahren
1 、 Konstruktion des prokaryotischen Expressionsplasmids: PCR des Zielgens, Vektorverdauung, Ligation, Transformation, Auswahl eines einzelnen Klons für die Sequenzierung;
2 、 verwandeln das erfolgreich konstruierte Plasmid in BL21 (DE3), inkubieren Sie über Nacht bei 37 ° C und wählen Sie den einzelnen Klon für ein kleines Schütteln über Nacht;
3 、 Kleine Induktion Um die besten Induktionsbedingungen zu finden: wird über Nacht die bakterielle Lösung 1: 1000 Inokulation in 3 ml lb, 37 ℃ 4-5h zu der Bakterienlösung OD600 = 0,6-0,8 Schütteln, hinzufügen, addieren unterschiedliche Konzentrationen von IPTG (0,1-1 mm), unterschiedliche Temperaturen, die 4-1-Inuktion der Temperatur, die Temperatur, die Temperatur, die Induktion der Temperatur. induziert 6H-8h, 16 ℃ induziert 16-20h, 16 ° C induziert 16-20h, die Temperatur wird verringert, die Zeit wird verlängert, z. C für 16-20h, nehmen Sie den bakteriellen Saft vor und nach der Induktion unter verschiedenen Induktionsbedingungen, führen Sie das Gel durch, flecken und beobachten Sie die Ergebnisse der Induktion; (Im Allgemeinen, je niedriger die IPTG -Konzentration und je niedriger die Induktionstemperatur ist, desto langsamer die Expression des Zielproteins, desto förderlicher für die korrekte Faltung von Proteinen, wodurch ihre Löslichkeit erhöht und die Erzeugung von Einschlusskörpern verringert wird)
4、After finding the most suitable induction conditions, inoculate the bacterial solution 1:1000 into 2L of LB, shake at 37℃ until the OD600 of the bacterial solution=0.6-0.8, aspirate 20μL of the bacterial solution and leave it to run the gel (1), then induce the protein expression under the suitable induction conditions obtained in the pre-experiment, and aspirate 20μL of the bacterial solution und lass es das Gel (2) laufen;
5 、 die induzierte bakterielle Flüssigkeit entfernen und 15 min bei 4 ℃ bei 4000 g zentrifizieren;
6 、 den Überstand verwerfen, wiegen, 10 ml Lysepuffer (1: 100 plus Proteaseinhibitor) für jede Gramm Bakterien, Resuspend, auf Eis für etwa 30 Minuten legen;
7 、 Druckquetscher: den Alkohol im Hochdruckhomogenisator ablegen, zweimal mit Wasser ausspülen, Lysepuffer einmal ausbalancieren, die bakterielle Flüssigkeit hinzufügen, Druck (Druck sollte 800 kPa nicht überschreiten), die bakterielle Flüssigkeit über drei- bis fünfmal bis transparent und nicht klebrig;
8 、 Sammeln Sie die zerkleinerte bakterielle Flüssigkeit, 12000 g, 4 ℃ Zentrifugation für 20 min, den Überstand und die Trennung von Niederschlag, wobei jeweils 20 & mgr; l Probe (3) (4) zurückgehalten wurde, um gelaufen zu werden;
9、2ml ni-nta wurde in die Reinigungssäule gegeben, um ethanol filtriert, mit Wasser gespült und Lysepuffer hinzugefügt, um die Säule zu äquilibrieren;
10 、 mit Lyse-Puffer Ni-nta Resuspension zum Überstand mischen gut, 4 ℃ Shaker-Inkubation für 2H;
11 、 Der Überstand wurde bei 4 ° C durch die Säule geleitet und 20 & mgr; l Filtrat wurden gesammelt (5);
12 、 Waschen Sie die Säule 3 mal mit 5 ml Waschpuffer und sammeln Sie die Filtratprobe 20 & mgr; l (6);
13 、 1 ml Elutionspuffer hinzufügen, 5 min inkubieren, den Eluat abholen, 5 mal wiederholen, 5 ml Eluat im selben Rohr sammeln, 20 μl Probe lassen (7);
14 、 Die im Verlauf des Experiments erhaltenen einzelnen Proteinproben wurden durchgeführt, mit Camumas brillant blau für 1 Stunden gefärbt und entfärbt, bis die blaue Hintergrundfarbe leichter war und klare Proteinbanden sichtbar waren;
15 、 Analysieren Sie die Proteinbanden jeder Probe gemäß den Ergebnissen der nachfolgenden Operation: Wenn die eluierten Proteinproben im Protein ohne heterozygote Banden oder heterozygote Banden oder weniger und flach sind, können nach der Diagryse und der Konzentration eine Dialyse und Konzentration wieder gereinigt werden.
16 、 Dialyse: Fügen Sie die Probe in die Dialysemembran hinzu, klemmen Sie beide Enden, Dialyse über Nacht bei 4 ℃;
17 、 Konzentration: Gemäß der Molekulargewichtsgröße der Probe zur Auswahl des geeigneten Konzentrationsröhrchens 4 ℃ Niedriggeschwindigkeitskonzentration, Konzentration der Proteinkonzentrationsmessung, Markierung, im flüssigen Stickstoff schnell und gefroren und dann in -80 ° C-Kühlschrank eingefroren.
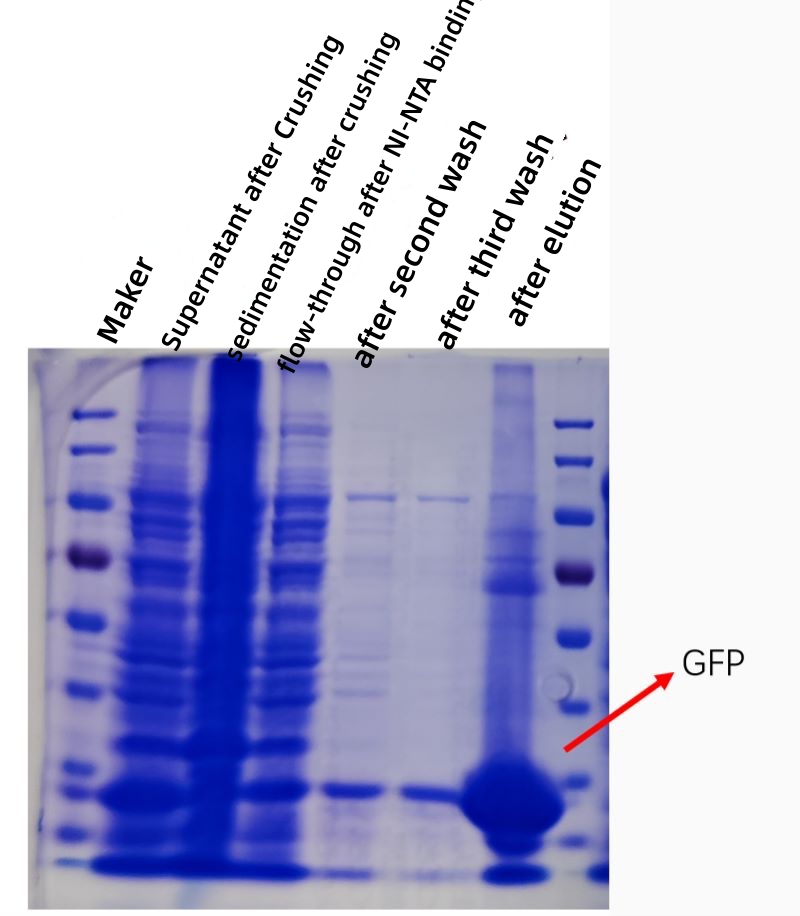
ERGEBNIS
Die folgende Abbildung zeigt ein Kofektionsdiagramm der GFP -Protein -Affinitätsreinigung in vitro, und die Reinheit und Konzentration des gereinigten Proteins ist stark erhöht