La purificación de proteínas es el proceso de introducción de secuencias de ácido nucleico que codifican las proteínas en las células huésped a través de técnicas de ingeniería genética, lo que hace que se expresen en grandes cantidades y luego las purguen in vitro utilizando métodos de purificación apropiados para obtener proteínas de alta pureza, actividad y rendimiento . Los sistemas de expresión de proteínas se pueden clasificar en sistemas procariotas y sistemas eucariotas.
Los principios y métodos para inducir la expresión de proteínas en sistemas procariotas y las proteínas purificadoras in vitro utilizando la cromatografía de afinidad se describen a continuación.
PRINCIPIO
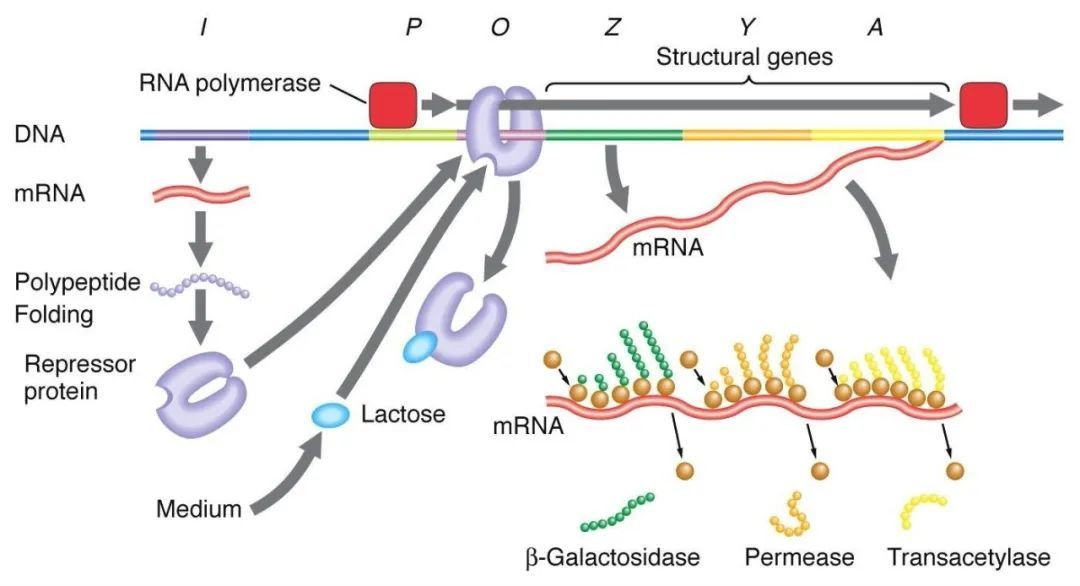
1 、 Regulación negativa del manipulador de lactosa:
En ausencia de lactosa, el manipulador LAC está en un estado de disuasión, momento en el que la secuencia I expresa la proteína disuasiva LAC para unirse a la secuencia O, evitando que la ARN polimerasa se una a la secuencia P e inhibe el inicio de la transcripción. Cuando la lactosa está presente, la lactosa ingresa a la célula y es catalizada por la β-galactosidasa y se convierte a la isolactosa, lo que se une a la proteína inhibitoria, lo que provoca un cambio conformacional en la proteína y conduce a la disociación de la proteína inhibitoria de la secuencia O, por lo tanto, inicia la transcripción.
El isopropiltiogalactosido (IPTG) actúa de la misma manera que la isogalactosa y es un inductor extremadamente potente que no es metabolizado por las bacterias y es muy estable y, por lo tanto, se usa ampliamente en los laboratorios.
2 、 Selección de tensión del huésped:
(1) BL21 es una de las cepas más utilizadas para la expresión procariota, que es principalmente adecuada para la expresión de proteínas no tóxicas con E. coli polimerasa, por lo que puede aplicarse a la expresión de sistemas procariotas que usan E. coli ARN polimerasa como TAC o TRC (EG PGEX, PLASMIDS PMAL).
(2) BL21 (DE3) integra el gen de ARN polimerasa de fago T7 en la región del fago λ DE3 en el cromosoma de la cepa BL21, que puede expresar tanto la ARN polimerasa T7 y la ARN polimerasa de E. coli , y puede usarse para la expresión de plásmidos como la serie PET, PGEX, PMAL y SO.
3, Métodos de purificación de proteínas:
(1) Cromatografía de filtración de gel: según el tamaño molecular de la mezcla a proteínas separadas, la forma de diferentes proteínas y el tamaño molecular de las diferencias en la mezcla a través de la columna de cromatografía de filtración en gel que contiene partículas de relleno, debido a la tamaño molecular de varias proteínas, el tamaño más grande es la diferencia de proteínas más grandes, la capacidad de la capacidad de la capacidad de la capacidad de la capacidad de la capacidad de las proteínas más grandes. ser eluyendo fuera de las moléculas son más pequeñas. Más tarde la elución.
(2) Cromatografía de intercambio iónico: la separación de proteínas y la purificación se basan en las diferentes cargas en la superficie de la proteína, la superficie de la proteína generalmente se carga de manera uniforme y se puede combinar con columnas de intercambio de cationes/aniones bajo ciertas condiciones. Cuando se cambia el pH o se usa un tampón con una resistencia iónica gradualmente que se usa para la elución, la sustancia unida se puede intercambiar con los iones en el eluyente y eluido en la solución. Dado que diferentes sustancias tienen diferentes cargas y diferentes habilidades de unión con la columna de intercambio iónico, el orden de ser eluyente en solución también es diferente.
(3) Cromatografía hidrofóbica: utilizando la hidrofobicidad de las proteínas, los residuos hidrofóbicos se expondrán en la superficie de las proteínas después de la desnaturalización o en un entorno de sal alto, los residuos hidrofóbicos de las diferentes proteínas tienen diferentes resistencias de acción con la resistencia hidrofóbica de la fase estacionaria, y la hidrofobicidad se puede usar como un componente más débil a la más débil de los componentes mediante la separación de los ligandos de los elementos de los elementos de los elementos de los elementos. bajo.
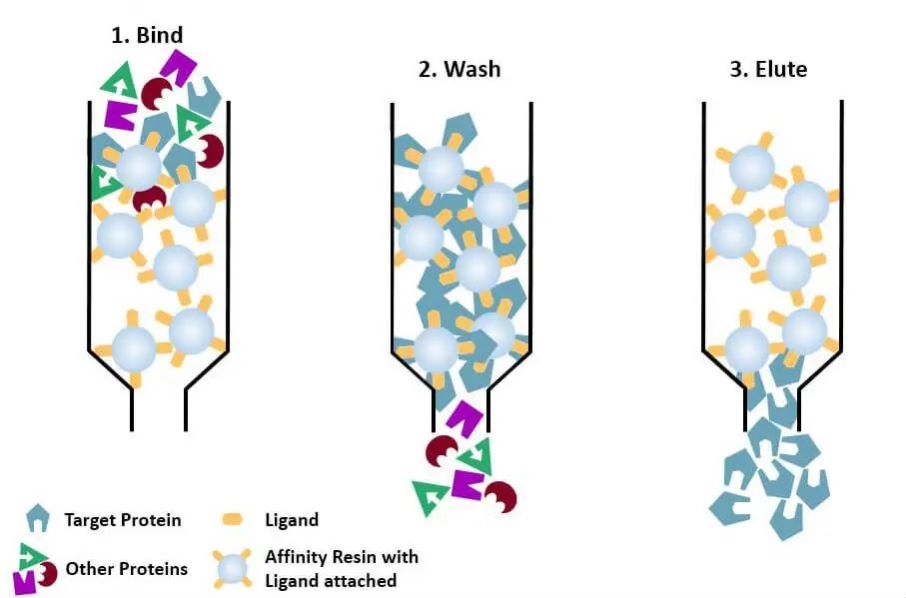
(4) Cromatografía de afinidad: el ligando específico de una proteína a purificar (o etiquetarse en la proteína) se une covalentemente a la molécula portadora mediante métodos químicos apropiados. Cuando la mezcla de proteínas se agrega a la columna cromatográfica llena con medio de afinidad, la proteína a purificar se une específicamente al ligando, mientras que las otras proteínas no están unidas y eliminadas mediante el lavado, y la proteína específicamente unida se puede eluir con una solución de ligando libre correspondiente. Las proteínas específicamente unidas se pueden eluir con una solución que contiene el ligando libre correspondiente.
4 、 Selección de etiquetas de purificación de proteínas:
Las características de las diferentes etiquetas son las siguientes ↓
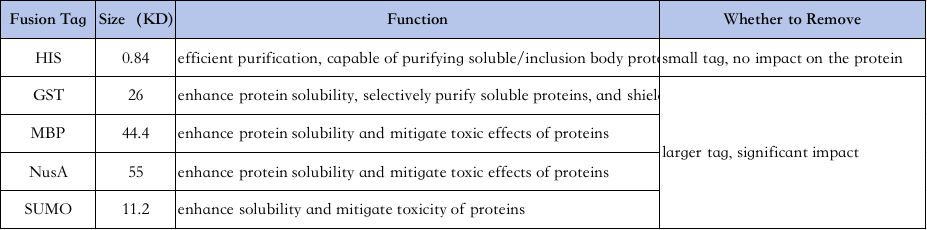
His-Tag Purificación: His-Tag es una de las etiquetas más utilizadas, donde se agregan 6-10 histidina al extremo amino o carboxilo de la proteína, y la proteína objetivo se purifica por su capacidad para unirse estrechamente a las columnas quelantes de Ni2+ en las condiciones normales o de desnaturalización (p. Ej., 8M URA), y luego se elee con imidazole (que compite con unión a los níquel o con unión normales o de unión).
Instrumentos y consumibles
Reactivos de laboratorio:
Iptg; Medio lb;
Tampón de lisis: Tris 20 mM (7.9), NaCl 500 mM, imidazol 10 mM;
Buffer de lavado: Tris 20 mM (7.9), NaCl 500 mM, imidazol 20 mM;
Buffer de elución: Tris de 20 mm (7.9), NaCl de 200 mm, 300 mm imidazol ( concentración de imidazol se puede ajustar de acuerdo con la eficiencia de elución );
Solución de diálisis: Tris 20 mM (7.9), NaCl 50 mM, 10% de glicerol;
Kaomas Solución brillante de tinción azul; Solución decolorante
Instrumentos:
Ni-NTA, interruptor ultrasónico, columna de cromatografía, tubos de concentración

Procedimiento
1 、 Construcción del plásmido de expresión procariota: PCR del gen objetivo, digestión vectorial, ligadura, transformación, recolección de clon único para la secuenciación;
2 、 Transforme el plásmido construido con éxito en BL21 (DE3), incubate a 37 ℃ durante la noche y elija el clon único para un pequeño agitación durante la noche;
3 、 Pequeña inducción Para descubrir las mejores condiciones de inducción: se agitará la solución bacteriana durante la noche 1: 1000 inoculación en 3 ml de lb, 37 ℃ agitando 4-5h a la solución bacteriana OD600 = 0.6-0.8, agregue diferentes concentraciones de IPTG (0.1-1mm), diferentes temperaturas, con la reducción de la temperatura, el tiempo de inducción se extiende, a medida que 37 ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃ ℃5 h. inducido 6H-8H, 16 ℃ inducido 16-20 h, 16 ° C inducido 16-20 h, la temperatura se reduce, el tiempo se extiende, como 37 ℃ inducido 4-5 h, 30 ℃ inducido 6H-8H, 16 ° C inducido 16-20 h, la temperatura se reduce. C para 16-20 h, tome la savia bacteriana antes y después de la inducción en diferentes condiciones de inducción, ejecute el gel, manche y observe los resultados de inducción; (En términos generales, cuanto menor sea la concentración de IPTG y cuanto menor sea la temperatura de inducción, más lenta es la expresión de la proteína objetivo, más propicio para el plegamiento correcto de las proteínas, aumentando así su solubilidad y reduciendo la generación de cuerpos de inclusión)
4 、 Después de encontrar las condiciones de inducción más adecuadas, inocular la solución bacteriana 1: 1000 en 2L de LB, agite a 37 ℃ hasta que el OD600 de la solución bacteriana = 0.6-0.8, aspirado 20 μl de la solución bacteriana y deje que ejecute el gel (1), luego induce la expresión de proteína bajo las condiciones de inducción adecuadas obtenidas en las condiciones de inducción adecuadas obtenidas en el pre-Experimento, y lo ejecute el gel de la tacción de gel. y deje que ejecute el gel (2);
5 、 Retire el líquido bacteriano inducido, centrífuga a 4000 g durante 15 minutos a 4 ℃;
6 、 Deseche el sobrenadante, pese, agregue 10 ml de tampón de lisis (1: 100 más inhibidor de la proteasa) para cada gramo de bacterias, resuspenda, coloque en hielo durante aproximadamente 30 minutos;
7 、 Atrolación de presión: posponga el alcohol en el homogeneizador de alta presión, enjuague con agua dos veces, use tampón de lisis para equilibrar una vez, agregue el líquido bacteriano, la presión (la presión no debe exceder los 800 kPa), el líquido bacteriano de tres a cinco veces a transparente y no pegajoso;
8 、 Recoja el líquido bacteriano triturado, 12000 g, 4 ℃ Centrifugación durante 20 minutos, la separación de sobrenadante y precipitado, cada una de las muestras de 20 μl retenidas (3) (4) que se ejecuta en gel;
Se añadieron 9、2ml Ni-NTA a la columna de purificación, para ser filtrado de etanol, enjuagado con agua y se agregó tampón de lisis para equilibrar la columna;
10 、 con tampón de lisis Ni-Nta resuspensión agregada al sobrenadante, mezclar bien, 4 ℃ incubación de agitador para 2h;
11 、 El sobrenadante se pasó a través de la columna a 4 ° C, y se recogieron 20 μl de filtrado (5);
12 、 Lave la columna con 5 ml de tampón de lavado 3 veces, recolecte la muestra de filtrado 20 μL (6);
13 、 Agregue 1 ml de amortiguador de elución, incubado durante 5 minutos, recolecte el eluato, repita 5 veces, recolecte 5 ml de eluato en el mismo tubo, deje 20 μl de muestra (7);
14 、 Las muestras de proteínas individuales obtenidas en el curso del experimento se ejecutaron, se tiñeron con azul brillante de color azul durante 1H y se decoloraron hasta que el color azul de fondo era más claro y las bandas de proteínas claras eran visibles;
15 、 Analice las bandas de proteínas de cada muestra, de acuerdo con los resultados de la operación posterior: si las muestras de proteínas eluidas en la proteína sin bandas heterocigotas o bandas heterocigotas o menos y poco profundas, pueden ser diálisis y concentración, si las bandas más heterocigosas deben purificarse nuevamente después de la diálisis y la concentración.
16 、 Diálisis: Agregue la muestra a la membrana de diálisis, sujete ambos extremos, diálisis a 4 ℃ durante la noche;
17 、 Concentración: según el tamaño de peso molecular de la muestra para elegir el tubo de concentración apropiado 4 ℃ Concentración de baja velocidad, concentración de medición de concentración de proteína, marcado, en la congelación rápida de nitrógeno líquido y luego congelado en -80 ℃ refrigerador.
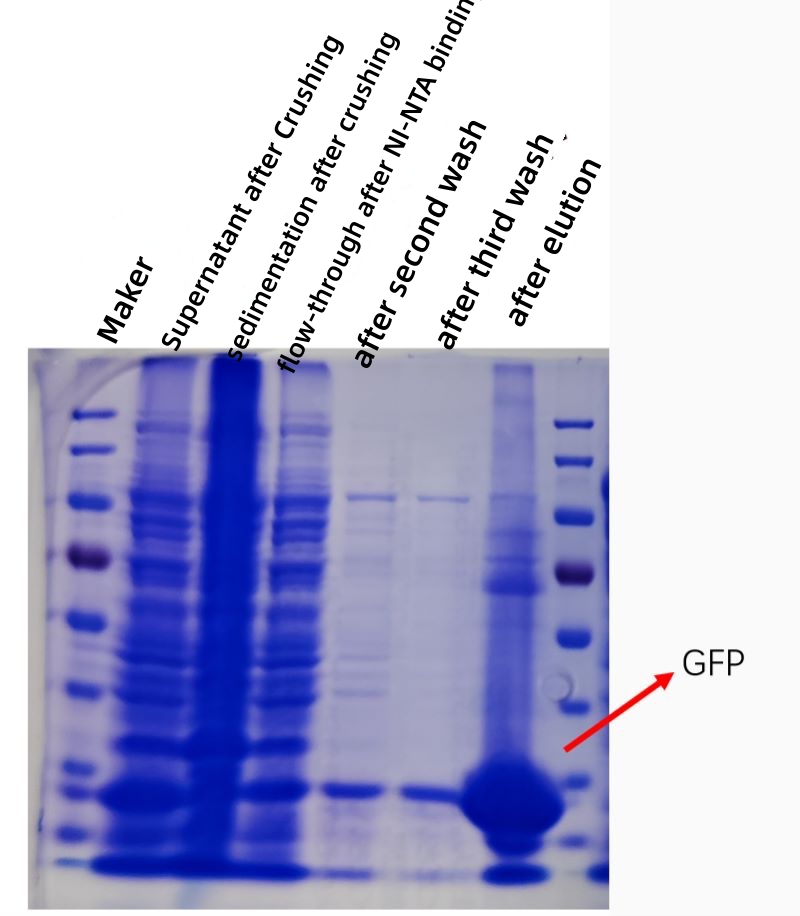
RESULTADO
La siguiente figura muestra una gráfica de kofección de la purificación de afinidad de la proteína GFP in vitro, y la pureza y la concentración de la proteína purificada aumentan considerablemente