La purificazione delle proteine è il processo di introduzione di sequenze di acido nucleico che codificano per le proteine nelle cellule ospiti attraverso tecniche di ingegneria genetica, causando loro espressi in grandi quantità e quindi purificandole in vitro usando metodi di purificazione appropriati per ottenere proteine di alta purezza, attività e resa . I sistemi di espressione proteica possono essere classificati in sistemi procariotici e sistemi eucariotici.
I principi e i metodi per indurre l'espressione proteica nei sistemi procariotici e le proteine purificanti in vitro usando la cromatografia di affinità sono descritti di seguito.
PRINCIPIO
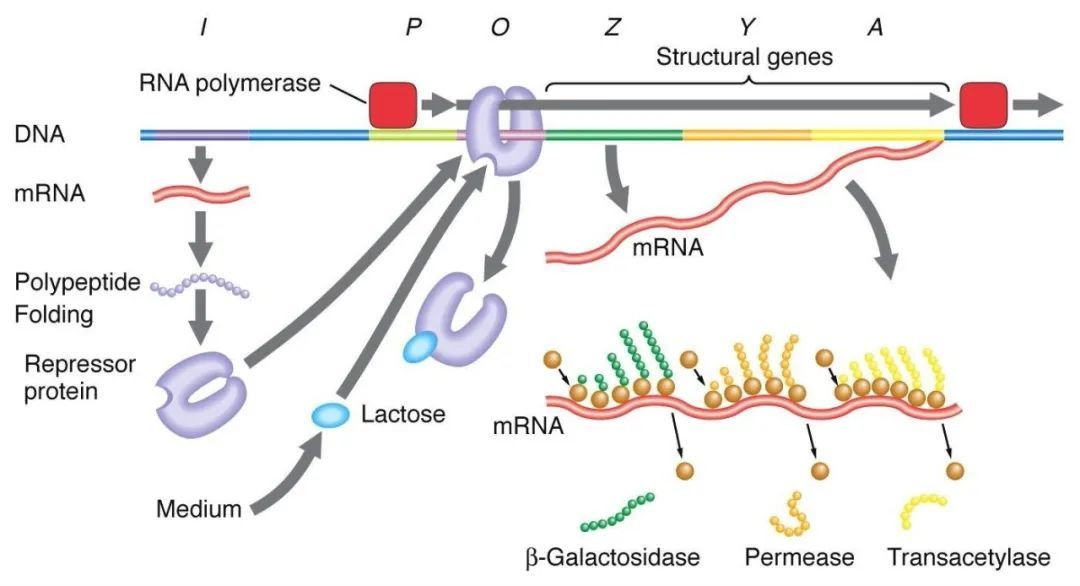
1 、 Regolazione negativa del manipolatore del lattosio:
In assenza di lattosio, il manipolatore LAC è in uno stato di deterrenza, quando la sequenza I esprime la proteina deterrente LAC per legarsi alla sequenza O, impedendo alla polimerasi dell'RNA di legarsi alla sequenza P e inibire l'inizio della trascrizione. Quando è presente il lattosio, il lattosio entra nella cellula ed è catalizzato dalla β-galattosidasi e convertito in isolactosio, che si lega alla proteina inibitoria, causando un cambiamento conformazionale nella proteina e portando alla dissociazione della proteina inibitoria dalla sequenza O, iniziando così la trascrizione.
L'isopropilthiogalattoside (IPTG) agisce allo stesso modo dell'isogalattosio ed è un induttore estremamente potente che non è metabolizzato dai batteri ed è molto stabile ed è quindi ampiamente utilizzato nei laboratori.
2 、 Selezione della deformazione host:
(1) BL21 è uno dei ceppi più comunemente usati per l'espressione procariotica, che è principalmente adatto all'espressione di proteine non tossiche con E. coli polimerasi, quindi può essere applicata all'espressione dei sistemi procariotici usando la polimerasi di RNA di E. coli come TAC o TRC (EG PGEX, PMALSIDS).
(2) BL21 (DE3) integra il gene T7 Phage RNA polimerasi nella regione del fago λ sul cromosoma del ceppo BL21, che può esprimere sia la polimerasi T7 RNA che la polimerasi di E. coli RNA e può essere usata per l'espressione di plasmidi come la serie PET, PGEX, PMAL e così.
3, Metodi di purificazione delle proteine:
(1) Cromatografia di filtrazione in gel: in base alla dimensione molecolare della miscela a proteine separate, alla forma di diverse proteine e alla dimensione molecolare delle differenze nella miscela attraverso la colonna di cromatografia di filtrazione del gel contenente le particelle di riempimento, a causa della dimensione molecolare delle dimensioni molecolari. Eluite dalle molecole sono più piccole, successivamente, successivamente l'eluizione.
(2) Cromatografia a scambio ionico: la separazione e la purificazione delle proteine si basano sulle diverse cariche sulla superficie della proteina, la superficie della proteina è generalmente caricata uniformemente e può essere combinata con colonne di scambio cation/anione in determinate condizioni. Quando il pH viene cambiato o un tampone con una resistenza ionica aumentata gradualmente viene utilizzato per l'eluizione, la sostanza legata può essere scambiata con gli ioni nell'eluente ed eluita nella soluzione. Poiché le diverse sostanze hanno cariche diverse e diverse capacità di legame con la colonna di scambio ionico, anche l'ordine di essere eluito in soluzione è diverso.
(3) Cromatografia idrofobica: l'uso dell'idrofobicità delle proteine, i residui idrofobici saranno esposti sulla superficie delle proteine dopo la denaturazione o in un ambiente salato elevato, i residui idrofobici di diverse proteine hanno una forza di azione idrofobica con la gradazione idrofobica e l'elua Basso.
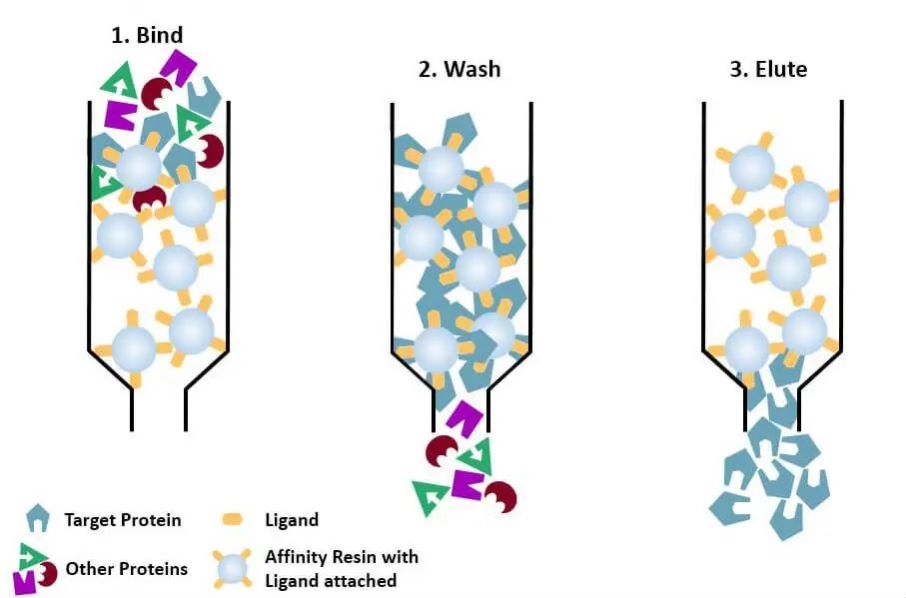
(4) Cromatografia di affinità: il ligando specifico di una proteina da purificare (o taggato sulla proteina) viene attaccato in modo covalente alla molecola portante con metodi chimici appropriati. Quando la miscela proteica viene aggiunta alla colonna cromatografica riempita con mezzo di affinità, la proteina da purificare è specificamente legata al ligando, mentre le altre proteine non sono legate e rimosse mediante il lavaggio e la proteina specificamente legata può essere eluita con una soluzione di ligando corrispondente libero. Le proteine specificamente legate possono essere eluite con una soluzione contenente il ligando corrispondente libero.
4 、 Selezione di etichette di purificazione delle proteine:
Le caratteristiche delle diverse etichette sono le seguenti ↓
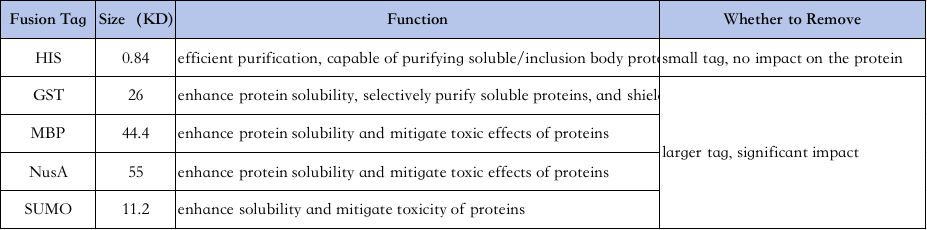
Purificazione del tag His: His-tag è uno dei tag più comunemente usati, in cui 6-10 istidina vengono aggiunte all'amino o al terminale carbossilico della proteina, e la proteina bersaglio viene purificata dalla sua capacità di legarsi strettamente a Ni2+ colonne chelanti nelle colonne di chelanti in condizioni di denunce in base alle colonne di chelanti in base alle colonne di chelanti in nickeling in nickeling in lesumi che hanno nickeling in base alle colonne di chelanti in base alle colonne di chelanti in nichelazioni nelle colonne di nickeling in nickeling in nickels in the BEads in zoti.
Strumenti e materiali di consumo
Reagenti di laboratorio:
Iptg; Medio LB;
Tampone di lisi: 20 mm Tris (7,9), NaCl 500 mm, imidazolo da 10 mm;
Tampone di lavaggio: 20 mm Tris (7,9), NaCl 500 mm, imidazolo da 20 mm;
Tampone di eluizione: 20mm Tris (7,9), NaCl 200mm, imidazolo da 300 mm ( la concentrazione di imidazolo può essere regolata in base all'efficienza di eluizione );
Soluzione di dialisi: Tris 20 mm (7,9), NaCl 50 mm, glicerolo al 10%;
Kaomas Brilliant Blue Staining Solution; Soluzione decolorante
Strumenti:
Ni-NTA, interruttore ad ultrasuoni, colonna di cromatografia, tubi di concentramento

Procedura
1 、 Costruzione del plasmide di espressione procariotica: PCR del gene bersaglio, digestione vettoriale, legatura, trasformazione, raccolta di clone singolo per il sequenziamento;
2 、 Trasformare il plasmide costruito con successo in BL21 (DE3), incubare a 37 ℃ durante la notte e scegliere il clone singolo per piccoli scuotimento durante la notte;
3 、 piccola induzione per scoprire le migliori condizioni di induzione: verrà scossa la soluzione batterica durante la notte 1: 1000 inoculazione in 3 ml di lb, 37 ℃ che scuotono 4-5 ore alla soluzione batterica OD600 = 0,6-0,8, aggiungi diverse concentrazioni di 4-5 di IPTG (0,1-1MM), diverse temperature, con la bassa temperatura, il tempo di induzione è esteso, come 37 ℃ a 4-5. 6h-8h indotto, 16 ℃ indotto 16-20 ore, 16 ° C indotto 16-20 ore, la temperatura è ridotta, il tempo è esteso, come 37 ℃ indotto 4-5h, 30 ℃ indotta 6h-8h, 16 ° C indotta 16-20 ore, la temperatura viene ridotta. C Per 16-20H, prendere la linfa batterica prima e dopo l'induzione in diverse condizioni di induzione, eseguire il gel, colorare e osservare i risultati dell'induzione; (In generale, minore è la concentrazione di IPTG e più bassa la temperatura di induzione, più lenta è l'espressione della proteina bersaglio, più favorevole al corretto piegatura delle proteine, aumentando così la loro solubilità e riducendo la generazione di corpi di inclusione)
4 、 Dopo aver trovato le condizioni di induzione più adatte, inoculare la soluzione batterica 1: 1000 in 2L di LB, agitare a 37 ℃ fino a quando l'OD600 della soluzione batterica = 0,6-0,8 e lascialo funzionare il gel (2);
5 、 Rimuovere il liquido batterico indotto, centrifuga a 4000 g per 15 minuti a 4 ℃;
6 、 scartare il surnatante, pesare, aggiungere tampone lisi da 10 ml (1: 100 più inibitore della proteasi) per ogni grammo di batteri, risospendere, posizionare il ghiaccio per circa 30 minuti;
7 、 Crushing a pressione: rimandare l'alcool nell'omogeneizzatore ad alta pressione, risciacquare con l'acqua due volte, usare il tampone di lisi per bilanciarsi una volta, aggiungere il liquido batterico, la pressurizzazione (la pressione non dovrebbe superare 800kpa), il liquido batterico su tre o cinque volte per trasparente e non appiccicoso;
8 、 Raccogliere il liquido batterico frantumato, 12000 g, 4 ℃ centrifugazione per 20 minuti, la separazione surnatante e precipitata, ciascuno mantenuto da 20 μl di campione (3) (4) da eseguire gel;
9、2ml Ni-NTA è stato aggiunto alla colonna di purificazione, per essere filtrato etanolo, sciacquato con acqua e aggiunto tampone di lisi per equilibrare la colonna;
10 、 Con tampone di lisi Ni-NTA Resuspensione aggiunta al surnatante, mescolare bene, 4 ℃ incubazione shaker per 2h;
11 、 Il surnatante è stato passato attraverso la colonna a 4 ° C e sono stati raccolti 20 μL di filtrato (5);
12 、 Lavare la colonna con tampone di lavaggio da 5 ml 3 volte, raccogliere il campione di filtrato 20 μl (6);
13 、 Aggiungere 1 ml di tampone di eluizione, incubare per 5 minuti, raccogliere l'eluato, ripetere 5 volte, raccogliere 5 ml di elui nello stesso tubo, lasciare 20 μl di campione (7);
14 、 I singoli campioni di proteine ottenuti nel corso dell'esperimento sono stati eseguiti, colorati con caumas brillante blu per 1H e decolorati fino a quando il colore blu di sfondo non era più chiaro e le bande proteiche chiare erano visibili;
15 、 Analizzare le bande proteiche di ciascun campione, secondo i risultati della successiva operazione: se la proteina eluita campioni nella proteina senza bande eterozigoti o bande eterozigoti o meno, possono essere dialisi e concentrazione, se più bande eterozigote devono essere purificate dopo la dialisi e la concentrazione.
16 、 Dialisi: aggiungere il campione nella membrana di dialisi, morsetto entrambe le estremità, dialisi a 4 ℃ durante la notte;
17 、 Concentrazione: in base alla dimensione del peso molecolare del campione per scegliere il tubo di concentrazione appropriato 4 ℃ concentrazione a bassa velocità, concentrazione della misurazione della concentrazione proteica, etichettatura, in congelamento rapido azoto liquido e quindi congelato in frigorifero -80 ℃ frigorifero.
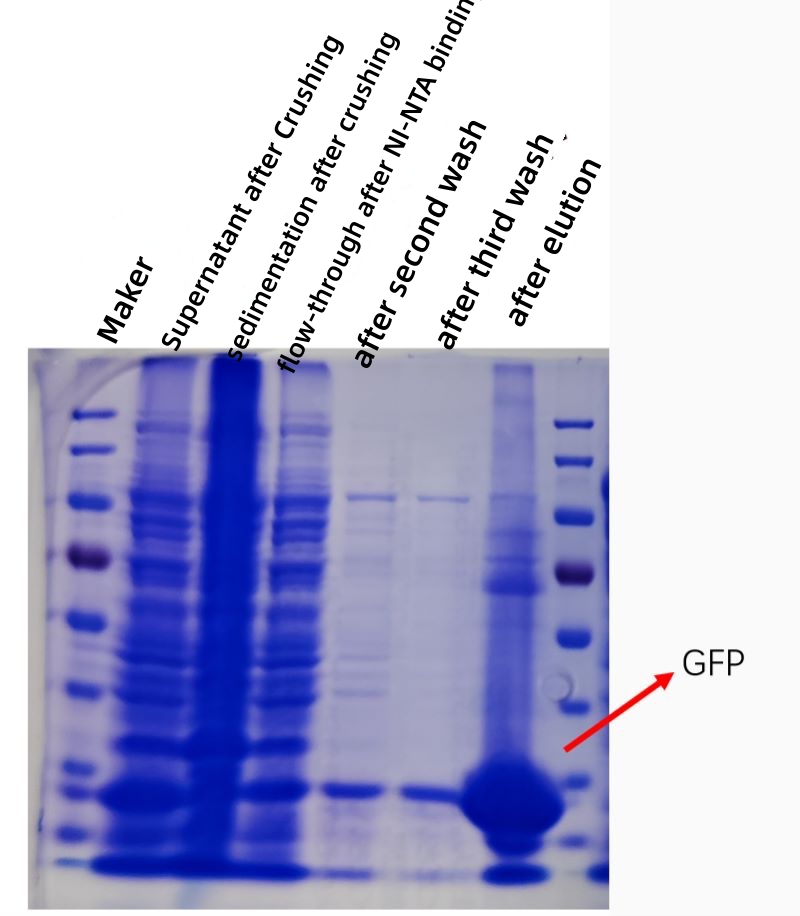
RISULTATO
La figura seguente mostra un diagramma di kofection della purificazione dell'affinità della proteina GFP in vitro e la purezza e la concentrazione della proteina purificata è notevolmente aumentata